10月3日,生物学领域重要期刊Life Science Alliance在线发表了37000cm威尼斯计智伟教授课题组的题为“scFseCluster: A Feature Selection Enhanced Clustering for Single Cell RNA-seq Data”的研究论文。针对同类算法普遍存在的泛化性能差或计算开销大等局限,他们开发了一种特征选择优化的单细胞聚类分析计算框架scFseCluster。

scFseCluster用于单细胞RNA -seq聚类分析主要包括两大步骤:首先,利用一种新开发的元启发式算法FSQSSA(基于量子松鼠搜索算法的特征选择)提取最优基因集;其次,基于最优基因集实现细胞聚类。研究人员选取了Goolam、Xin、Montoro、Hrvatin等8个基准数据集(细胞数目从100到50000不等),对算法进行了全面测试。实验结果表明,scFseCluster在所有数据集上均表现卓越,预测性能明显优于其他七种主流算法,包括Seurat(Nat Biotech, 2015)、CIDR(Genome Biol, 2017)、SINCERA(PlosComput Biol, 2015)、SIMLR(Nat Methods, 2017)、DESC(Nat Commun, 2020)、SC3(Nat Methods, 2017)、scDeepCluster(Nat Mach Intell, 2019)。总之,scFseCluster在各种尺度的单细胞RNA-seq数据集上均表现出较高的聚类性能。这项研究建议:在对scRNA-seq作聚类分析时应更多关注基因选择,降低对聚类模型的依赖。
研究人员首先证明了FSQSSA用于基因选择的优越性能。他们在上述八个公共数据集上对比了FSQSSA与四种群智能搜索算法分别用于基因选择的性能差异。从图1可见,FSQSSA算法获得的最优基因集的适应度值明显优于其他四种算法(图1A)。此外,FSQSSA 还表现出极快的收敛速度,能以很少的迭代次数收敛到最优基因集(图1B)。

图1. FSQSSA与其他群智能搜索算法在基因选择方面的性能比较
研究人员在所有数据集上测试了FSQSSA算法的鲁棒性。FSQSSA在每个数据集上均被独立重复500次,以评估算法最优解的变化。从收敛曲线可以看出,除了数据集Romanov以外,FSQSSA在其他七个数据集上的最优解变化很小,表明种群随机初始化并没有对算法收敛性能产生显著影响(见原文图2)。
为了研究算法在细胞聚类时的性能,研究者将scFseCluster应用于上述8个已标记细胞类型的scRNA-seq数据集。采用了六个指标(ARI、RI、AMI、NMI、ACC、和FMI)来评价聚类结果的优劣。如下图2所示,scFseCluster在所有数据集上均表现出色,尤其是数据集Xin,Goolam,Usoskin和Hrvatin。对比分析显示,scFseCluster在六个评价指标上明显优于其他7种现有方法。
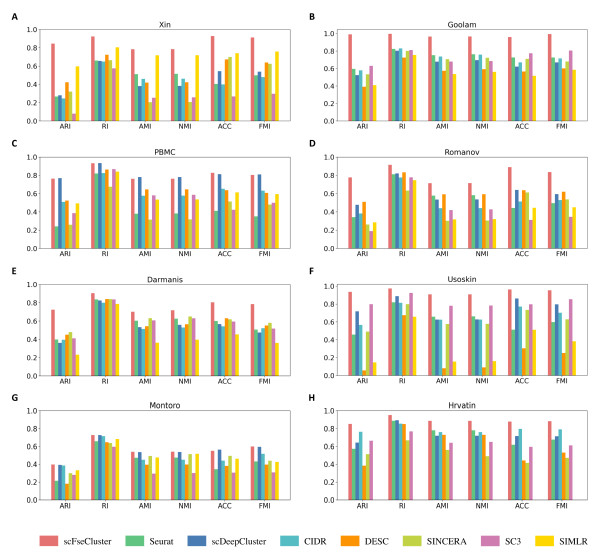
图2. scFseCluster与其他7中SOTA方法的聚类性能对比
此外,研究人员采用t-SNE在二维空间对细胞聚类结果进行可视化展示。从图3可见,scFseCluster在不同尺度样本量的数据集上均表现良好。平均而言,scFseCluster在八个数据集上的细胞聚类准确率高达85.05%,明显优于其他方法。

图3. 八种算法对单细胞RNA-seq聚类分析的可视化结果
当前流行的单细胞RNA-seq聚类方法常用2000个高变基因作为输入,再通过各种不同的计算策略实现细胞聚类。scFseCluster则通过特征选择策略先选取最优基因集,再采用最为常见的K-means算法就能实现高精度细胞聚类。其特点是尽量简化聚类任务,而把重心放在对最优基因集选取的优化上。原文图5展示了FSQSSA算法可进一步提升现有SOTA方法的聚类性能。通过引入FSQSSA算法作为预处理步骤,使得前六种SOTA 方法的ARI 和ACC值平均提高0.11和0.09。特别地,当FSQSSA结合了Seurat以后,可使其ARI值从0.24增加到0.75,ACC值从0.41提升至到0.81。综上所述,研究证明了FSQSSA对于提升单细胞RNA-seq聚类算法性能的广泛适用性。
综上所述,该研究提出了一种用特征选择策略优化的单细胞RNA-seq聚类分析框架scFseCluster。研究结果证实,scFseCluster适用于多种尺度的单细胞RNA-seq数据集,并表现出很高的聚类精度和鲁棒性。更重要的是,这项研究认为“目前较为流行的方法通过选取2000个高变基因来执行细胞聚类”的计算策略存在一定局限性,并强调了基因选择对于聚类分析的重要性。此外,研究人员也发现,scFseCluster模型在处理细胞数目超过1万以上的大规模数据集时,计算性能并不占优势。为此,他们近期还成功开发了一款基于基因排序策略的单细胞RNA-seq快速聚类算法FRSC,相信在不久的将来就会问世。
本文的第一作者为37000cm威尼斯已毕业专硕王宗钦,通讯作者为计智伟教授。UNC Chap Hill的Weiling Zhao教授为文章的撰写提供了宝贵意见。该成果获得南京农业大学海外高层次引进人才启动项目、江苏省自然科学基金项目、江苏省农业科技自主创新项目、科技部外专项目、中央高校基本业务经费等项目的支持。
原文链接:https://doi.org/10.26508/lsa.202302103